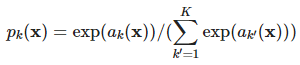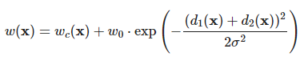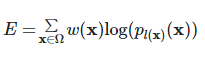이 글은 U-Net: Convolutional Networks for Biomedical Image Segmentation 논문을 읽고 정리한 것입니다. 이 글에 있는 대부분의 사진들은 논문에서 가져온 것임을 밝힙니다.
Abstract
- 본 논문에서는 적은 데이터셋을 더 효율적으로 사용하기 위한 data augmentation 기법과 이를 통해 학습한 네트워크(U-Net)를 제안한다.
- U-Net에 대해 정리하면 다음과 같다.
- Contracting path와 expanding path로 구성된다.
- 적은 학습 이미지로부터 end-to-end로 학습한다.
- ISBI challenge에서 가장 높은 성능을 기록하였다.
Introduction
- Biomedical image processing 분야의 특징은 다음과 같다.
- 출력 결과는 반드시 localization을 포함해야 하므로, 각 픽셀 별 classification(segmentation)을 수행해야 한다.
- 일반적으로 biomedical task에서는 수천개의 이미지 데이터를 구축하기 어려우며, 이는 학습에 사용가능한 이미지의 수가 적다는 것을 의미한다.
- ISBI 2012의 EM segmentation challenge에서 가장 높은 성능을 기록하였던 Ciresen et al.의 연구에는 다음의 2가지 단점이 존재하였다.
- 이미지로부터 patch를 추출하여 학습하는 방법을 사용하였는데, 네트워크가 각 patch별로 독립적으로 동작하기 때문에 속도가 느렸고 겹치는 patch들로 인한 중복(redundancy)이 많았다.
- Localization accuracy와 네트워크가 바라보는 context간의 trade-off가 있었다.
- Large patch –> more max-pooling layer –> reduce localization accuracy
- Small patch –> less max-pooling layer –> see only little context
- 본 논문에서는 다음과 같이 fully convolutional network(FCN)를 수정하여 적은 학습 이미지로 정확한 segmentation을 수행할 수 있는 U-Net architecture를 제안한다.
- Upsampling에서 더 많은 feature channel을 사용하여 네트워크가 context 정보를 상위 layer로 전파할 수 있도록 하였다.
- Contracting path에서의 feature를 expanding path로 copy and crop
- Fully connected를 사용하지 않고 convolution만을 사용하였다.
- Overlap-tile strategy를 통해 patch단위로 segmentation을 수행하여 큰 이미지에 대해서도 중간에 끊김 없이 매끄러운 segmentation 결과를 얻을 수 있도록 하였다.
- 이미지의 테두리에서는 mirroring을 통해 missing context를 보완하였다.
- 큰 이미지에서 GPU 메모리의 부족으로 인해 해상도가 제한되는 것을 막아준다.
- Elastic deformation을 적용한 data augmentation을 수행하였다. Read more relating to online-casino suomi.
- 부족한 학습 데이터를 보완하였다.
- 네트워크가 deformation에서 robust하도록 해준다.
- 세포 조직이 각 순간순간 마다 조금씩 변화하는 것을 elastic deformation이 효과적으로 시뮬레이션 할 수 있기 때문에, biomedical segmentation에서는 매우 효과적이다.
- 같은 class이면서 가까이 붙어있는 cell들을 분리(instance segementation)하기 위해 weighted loss를 사용하였다.
- Loss function에서 붙어있는 cell들 사이에서 이들을 분리하는 border의 background label이 큰 weight를 갖도록 하였다.
- U-Net은 다양한 biomedical segmentation에 적용할 수 있다.
- ISBI의 EM segmentation challenge와 cell tracking challenge에서 가장 높은 성능을 기록하였다.
Network Architecture

- U-Net architecture는 다음과 같이 구성된다. (Figure 1)
- Contracting path (downsampling)
- 3××3 convolution (padding X), batch normalization, ReLU
- 2×2 max pooling (stride 2)
- conv, max pool을 통한 각 downsampling step마다 output feature channel의 수는 2배가 된다.
- Expansive path (upsampling)
- 2××2 up-convolution (transposed convolution)
- Feature channel을 반으로 줄이고, contracting path의 feature map을 concatenate(copy and crop)
- 3××3 convolution (padding X), batch normalization, ReLU
- Final layer (output map)
- Over-lap tiling을 적용하기 위해서는 2××2 max pooling으로 최종 출력 크기가 원본 크기와 같아지는 input tile size를 선택해야 한다.
Training

- 네트워크의 학습에는 Caffe 프레임워크의 stochasic gradient descent를 사용하였다.
- Unpadded convolution으로 인해 출력 이미지는 입력 이미지보다 크기가 줄어든다. (Figure 2)
- Over-lap tiling을 위해 원본 이미지에 mirroring으로 테두리를 추가한 이미지가 입력으로 들어가게 되고, 추가된 테두리만큼 출력 이미지의 크기는 줄어들게 된다.
- GPU 메모리 사용을 최대로 하기 위해 batch size보다 input tile size를 크게하는 방법을 사용하였다.
- Batch size는 1(single image)로 하였다.
- 작은 batch size를 보완하기 위해 momentum은 0.99로 큰 값을 사용하여 previous step에서의 여러 training sample들이 current step에서의 update에 더 많은 영향을 미치도록 하였다.

- Energy function은 최종 feature map에서의 pixel-wise softmax와 weight map에 cross entropy loss를 적용한 형태이다.
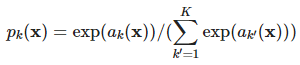
-
-
- ak(x)ak(x)은 픽셀 위치 x∈Ωx∈Ω에서 feature channel kk의 activation이다.
- 즉, pk(x)≈1pk(x)≈1이라면, k번째 channel에서 activation(ak(x)ak(x))이 가장 커서 kk의 확률이 가장 높다는 의미이다.
- Weight map w(x)w(x) (Figure 3)
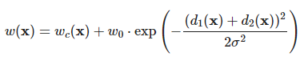
-
-
- Weight map은 학습 데이터에서의 class간 imbalance를 맞춰주고, 가까이 붙어있는 cell들 사이의 작은 경계(border)를 학습할 수 있도록 하기 위해 미리 계산하는 값이다.
- wc:Ω→Rwc:Ω→R은 class frequency의 균형을 맞춰주기 위한 weight map이다.
- d1:Ω→Rd1:Ω→R은 가장 가까운 cell의 경계(border)까지의 거리이다.
- d2:Ω→Rd2:Ω→R은 두번째로 가까운 cell의 경계(border)까지의 거리이다.
- 논문에서는 w0=10,σ≈5w0=10,σ≈5로 두고 실험을 수행하였다.
- pk(x)pk(x)와 w(x)w(x)에 cross entropy를 적용한 energy function EE
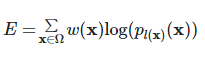
-
-
- Energy function은 각 픽셀 위치에서 pl(x)(x)pl(x)(x)와 1의 차이에 페널티를 준다.
- 즉, 정답일 확률이 낮을수록 패널티를 주어 정답 확률이 높아지도록 하는 것이다.
- l:Ω→1,⋯,Kl:Ω→1,⋯,K은 각 픽셀에 대한 true label을 의미한다.
- 즉, pl(x)(x)pl(x)(x)는 정답 label(k=lk=l)에서의 softmax 결과(정답의 확률)를 의미한다.
- Weight Initialization에는 He Initialization을 사용하였다.
Data Augmentation
 그림 출처 : Towards Data Science – Review: U-Net (Biomedical Image Segmentation)
그림 출처 : Towards Data Science – Review: U-Net (Biomedical Image Segmentation)
- Random elastic deformation을 통해 data augmentation을 수행하였으며, 이는 적은 데이터로도 네트워크를 학습할 수 있었던 key concept이다.
- Microscopical image의 경우, shift and rotation invariance와 robustness to deformation and gray value variations가 요구되는데 이를 충족시켰다.
Experiments



- U-Net은 3가지의 다른 segmentation task에서 모두 좋은 성능을 보였다.
- EM segmentation challenge
- Cell tracking challenge의 2가지 dataset
- Dataset(“PhC-U373”)은 Figure 4 – (a), (b)를 참고
- Dataset(“DIC-HeLa”)은 Figure 3, Figure 4 – (c), (d)를 참고
Conclusion
- U-Net은 biomedical segmentation task에서 높은 성능을 기록하였다.
- Elastic deformation을 통한 data augmentation을 통해 적은 이미지 데이터로 네트워크를 성공적으로 학습할 수 있었다.
출처 : https://younnggsuk.github.io/2021/02/08/u-net_convolutional_networks_for_biomedical_image_segmentation.html